如果只有几个位点,好说,可以直接上NCBI一个一个查,如果位点一多,就需要批量查询了!
第一种方法: 使用annovar进行注释。 annovar提供了一个把rsid注释出region location的方法:
convert2annovar.pl -format rsid rsid.txt -dbsnpfile humandb/hg19_snp138.txt > tempfile
table_annovar.pl tempfile humandb/ \
-buildver hg19 \
-out rsid.anno \
-remove \
-protocol refGene,cytoBand,snp138,avsnp150 \
-operation g,r,f,f \
-nastring . \
-thread 10 \
-otherinfo
rm tempfile
然而annovar目前使用的dbsnp版本太旧,很多位点无法注释出来,所以这个方法不怎么好。
第二种方法:
使用NCBI提供的网页工具rslist。
操作上还是挺简单的。不过,必须提供邮箱来接收结果,而且,NCBI在今年(2018)六月就会关闭这个工具。
第三种方法:
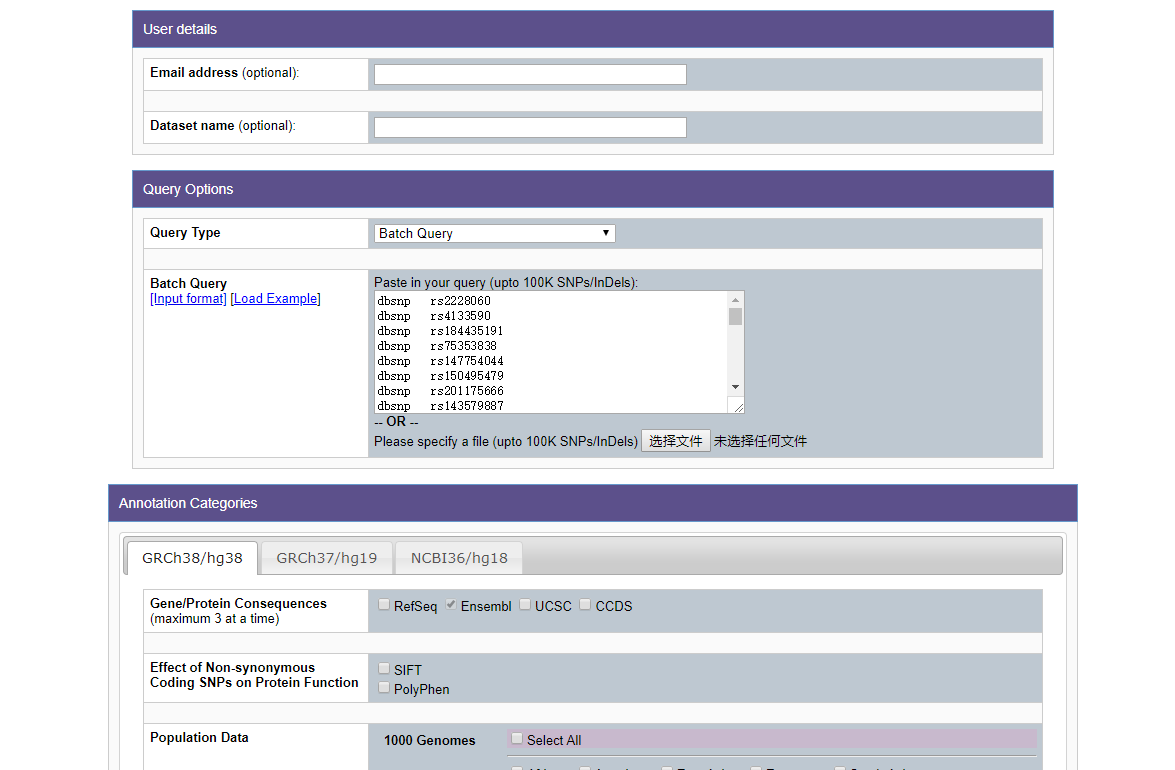
是我目前觉得最好用的,也是一个网页工具:snp-nexus
能进行注释的内容还是很多的,如图这样输入rsid。选择要注释的数据库以及参考基因版本。
我建议无论是做37还是38的都选择38版本。 因为有部分新的rsid好像在37版本的dbsnp里没有! 先把38的位置注释出来,再利用ucsc的lift over把坐标置换成37就好了。(虽然不知道可不可以这样操作。。) snp nexus里也可以选择发送到邮件,也可以不选。 等几分钟结果就会出来(看注释多少个位点和数据库)。
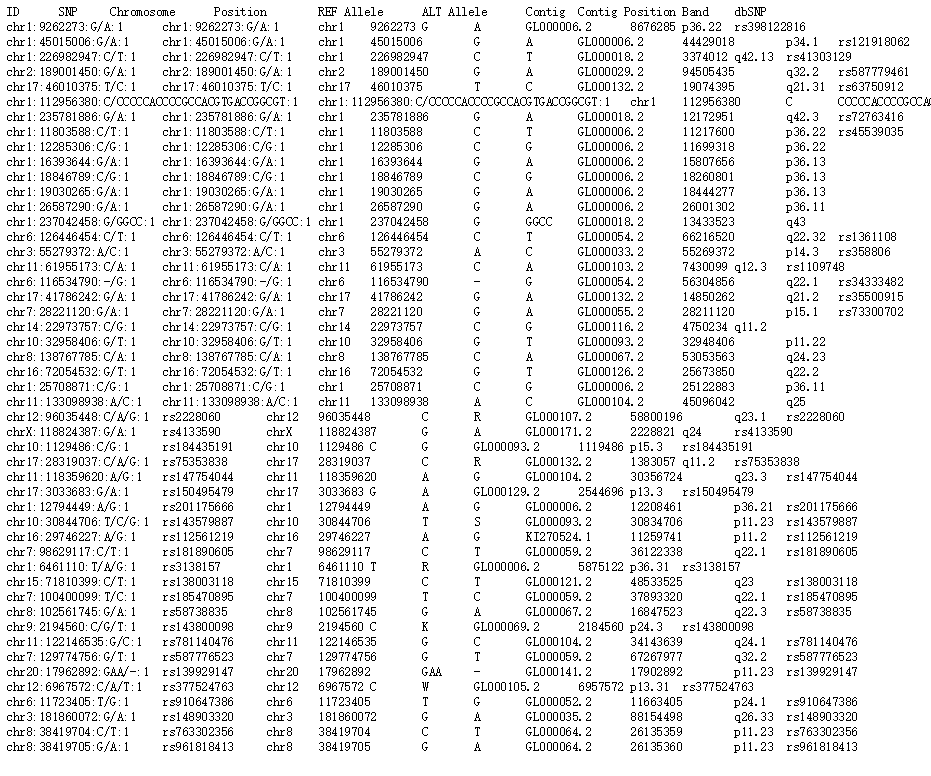
一个默认获得的结果是这样的:
随便撸个脚本处理一下就美滋滋了!