用clusterProfiler已经能出很好的图了。但是有时需要满足别的需求。
下载模拟的基因列表,也可以用自己的。
wget https://raw.githubusercontent.com/pzweuj/practice/master/R/GO_KEGG/gene.list
然后下面以GO CC注释为例
library(org.Hs.eg.db)
library(clusterProfiler)
library(ggplot2)
# 读入文件
data <- read.table("gene.list",header=TRUE)
data$GeneName <- as.character(data$GeneName)
transID = bitr(data$GeneName,
fromType="SYMBOL",
toType=c("ENSEMBL", "ENTREZID"),
OrgDb="org.Hs.eg.db"
)
# 富集分析
CC <- enrichGO(transID$ENTREZID,
"org.Hs.eg.db",
keyType="ENTREZID",
ont="CC",
pvalueCutoff=0.05,
pAdjustMethod="BH",
qvalueCutoff=0.1
)
CC <- setReadable(CC, OrgDb=org.Hs.eg.db)
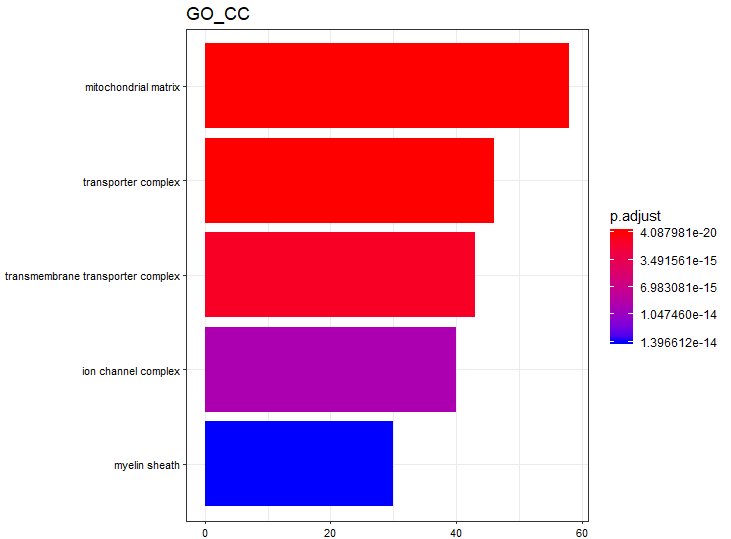
# 一般的,可以这样画
barplot(CC, showCategory=5, title="GO_CC", font.size=8)
结果是这样的
输出excel结果。
# 输出结果
CC_df <- as.data.frame(CC@result)
write.table(CC_df, file="GO_CC.xls", sep="\t", row.names=F)
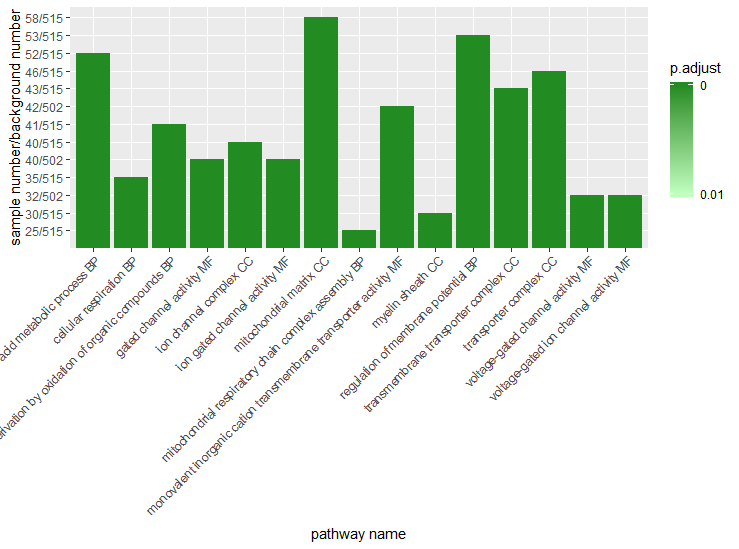
然后这是用ggplot2画图
# 太多的话不好输出,这里只取前5个(按p值排列)
CC_top5 <- head(CC_df, n=5)
# 这次就是用ggplot2画图
p <- ggplot(CC_top5, aes(x=CC_top5$Description, y=CC_top5$BgRatio, fill=CC_top5$p.adjust)) + geom_bar(stat="identity") +
theme(axis.text.x = element_text(angle = 45, hjust = 0.5, vjust = 0.5)) +
ylab("sample number/background number") +
xlab("pathway name") +
labs(fill="p.adjust") +
scale_fill_gradient(low="#C1FFC1", high="#228B22")
p
结果如下
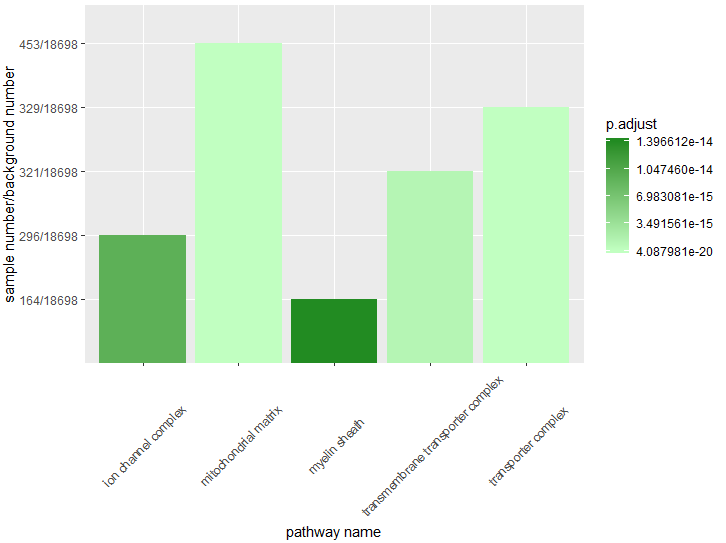
##20180905更新 p值越小,置信等级越高,所以这里需要把图例反过来。
采取一个投机取巧的方法,把p值求相反数。
CC_top5$p.adjust2 <- 0 - CC_top5$p.adjust
然后再来画图,注意这里的labels和breaks是一一对应的。labels是图例显示,而breaks是实际中的断点。
p <- ggplot(CC_top5, aes(x=CC_top5$Description, y=CC_top5$GeneRatio, fill=CC_top5$p.adjust2)) + geom_bar(stat="identity") +
theme(axis.text.x = element_text(angle = 45, hjust = 1, vjust = 1)) +
ylab("sample number/background number") +
xlab("pathway name") +
labs(fill="p.adjust") +
scale_fill_gradient(low="#C1FFC1",
high="#228B22",
labels=c("0.01", "0"),
breaks=c(-0.01, 0),
limits=c(-0.01, 0)
)
p