很多时候,看文献啊报告这类东西的时候作者因为某些原因,不会给出明确位点,只会给基因名和碱基突变或蛋白质突变。例如,给出 ITPA c.94C>A 这样的突变。
这里介绍一个可以凭这点信息找到绝对位置(染色体+物理长度)的工具:Transvar
Transvar是一个用python写的开源软件,我们可以下载下来使用,但是下载的话还要下载数据库比较麻烦。所以可以选择使用网页版。
网页版还是一个做了用户界面的,非常贴心而且非常容易使用。看一眼就会的那种。
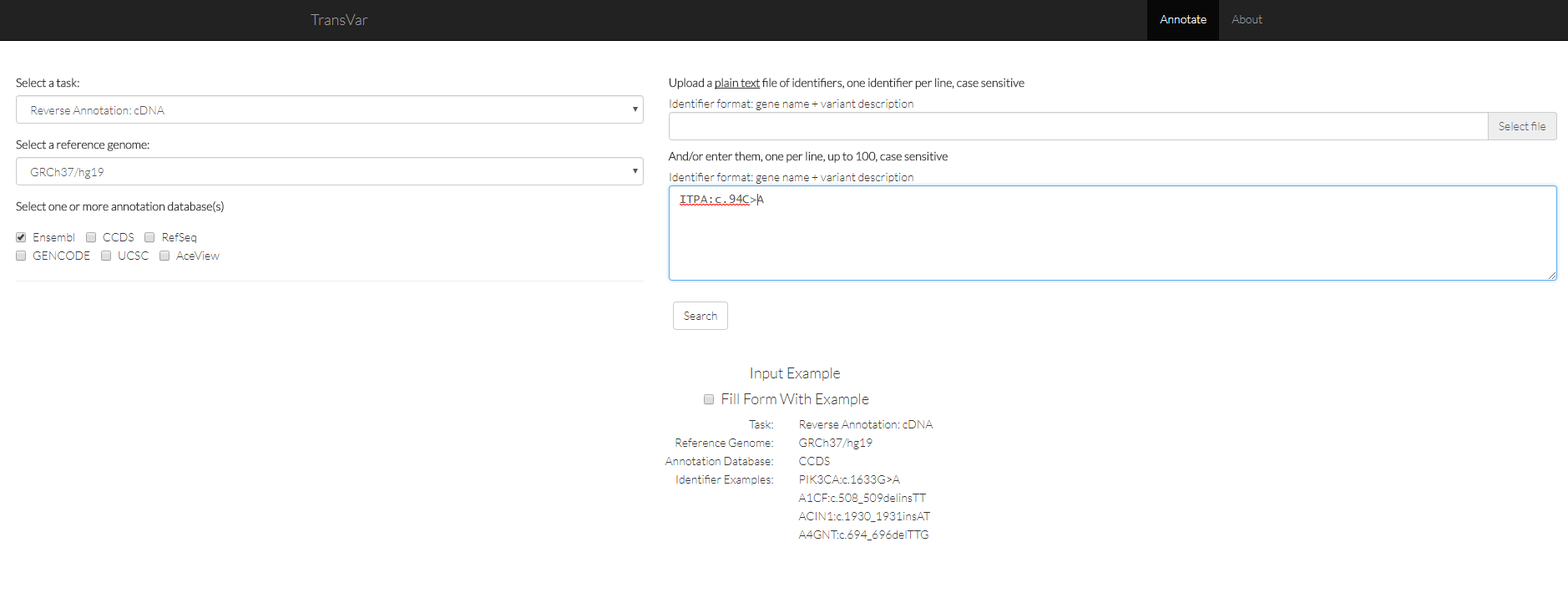
Upload a plain text顾名思义是可以上传纯文本。注意文本格式要按照下面提供的example格式。一行只能有一个突变。
而下面的框框则是可以不上传文本而是直接输入。格式也要照着下面,而且也只能一行一个,最多100行。
填好之后,点击search。
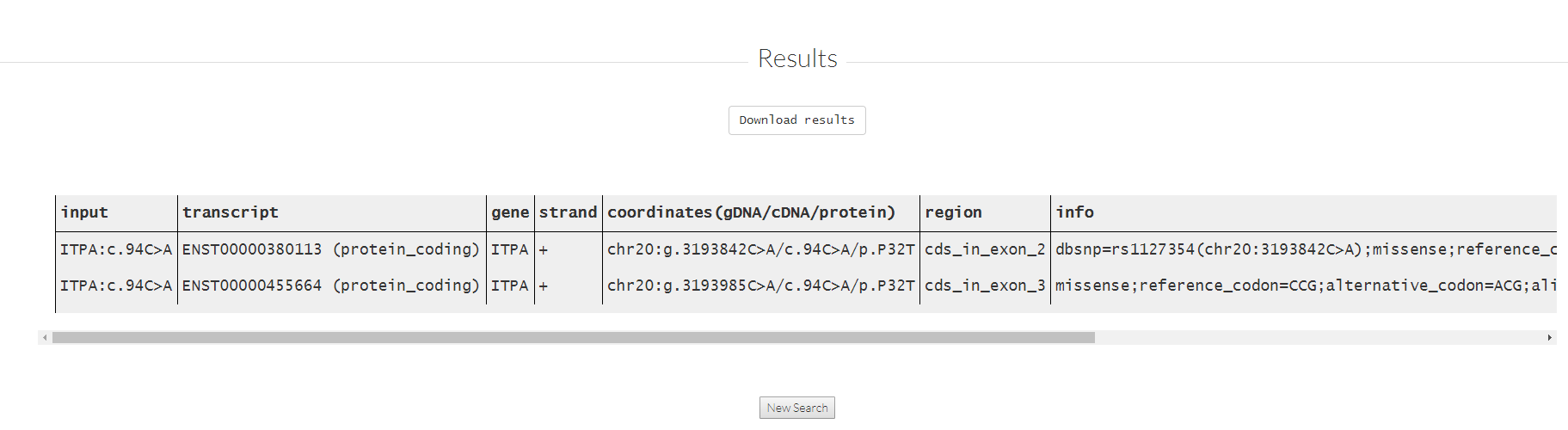
得到结果。
我们可以复制结果,也可以点击Download results下载结果。