简单的,做panel的,例子
panel就是关于某个疾病的位点,一般包括这个疾病的致病基因位点,药敏基因位点,毒副作用基因位点,风险基因位点这些。 很多时候都要靠手工去查询文献来手动添加。或者,可以去购买商业化的疾病panel。
一个panel,最重要的是可信。可信的意思是,位点要有相关文献的支持。 然后位点要的是参考基因组上的绝对位置。 例如hg19的chr1:0000001这样的。
下面利用这个公开的老年痴呆数据库来建立一个panel。
这个数据库的有点在于,每个位点都有pmid,而且标注了位点是致病的还是良性的。 首先,我们通过浏览器右键查看源代码简单粗暴的把这个网页弄下来。
当然,页面很多的情况下是写一个爬虫程序来把网页弄下来,但是我们需要的只是一个页面,显然不需要这么做。
点这里查看网页的源代码。
接下来就是写脚本来处理这个网页的源代码,把需要的信息提取出来。 下面是我用来处理的python脚本:
from bs4 import BeautifulSoup
inputFile = open('AD.html', 'r')
outputFile = open('result.txt', 'w')
soup = BeautifulSoup(inputFile, 'lxml')
gene = soup.select('p')
genes = []
for i in range(len(gene)):
if str(gene[i]).__contains__('span class'):
genes.append(gene[i])
genes.pop()
geneall = []
for m in range(len(genes)):
l = str(genes[m]).split('\n')
line = ''.join(l)
lines = ''.join(line.split())
genelist = lines.split('<b><i>')
del genelist[0]
genelist[-1] = genelist[-1].replace('</p>', '')
for n in range(len(genelist)):
geneall.append(genelist[n])
for x in geneall:
genename = x.split('</i></b>:')[0]
aachange = x.split('</i></b>:')[1]
aachanges = aachange.split('span')
del aachanges[0]
del aachanges[-1]
aachangess = ''.join(aachanges).split('>)(<')
for item in aachangess:
genedes = genename + '\t' + item.split('">')[0].split('"')[1] + '\t' + item.split('">')[1].split('<')[0]
outputFile.write(genedes + '\n')
outputFile.close()
inputFile.close()
print 'task done'
处理完之后,是这样的:
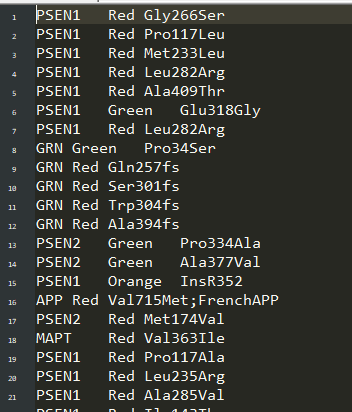
然后把它弄到excel中,用excel处理。 我个人的想法是,能简单用excel处理就行的东西,不要浪费时间写代码处理。
其中red是致病的,我们用excel把这些筛选处理就好了。 把基因和aachange提取出来之后,可以去Transvar 进行反注释。
注释之后的结果,利用excel进行筛选。 利用left(),right(),提取染色体+物理位置这种绝对路径。
然后把这些位置弄出来,格式大概是这样:
{plain text}
#CHROM POS ID REF ALT
1 12312312 AD_1 C T . . .
需要8列,前面五列这样就ok,后面可以用点点点代替。
拿去用annovar注释,得到多种信息。
这样就是一个简单的panel了。