自从知道这款软件,工作效率提高了不少。
这是一款输入表型和下机的vcf就可以输出与表型最相关的基因的软件。目前看来效果很好。用java写的,注意系统要有java的环境。 首先例行放上软件的主页。 Exomiser
然后这里是下载地址。 Installation windows、linux、mac版本都有,按自己的系统安装就行了。
记得还要下载它的数据库。 data
简单的操作介绍,简直是官网的典范。 manual
接下来讲一下基本操作。 首先,得到患者的临床信息,把表型信息分析出来,去获得表型的HP号。所谓的HP号,就是Human Phenotype Ontology对表型的编号。 中文的话,可以去奇恩生物的罕见病辅助诊断系统。 英文的话,就去HPO提供的phenomizer。
比如说,我知道患者有两个表型,一个是全面强直阵挛,一个是癫痫持续状态。
搜索后,可以得到HP号。HP:0002069。
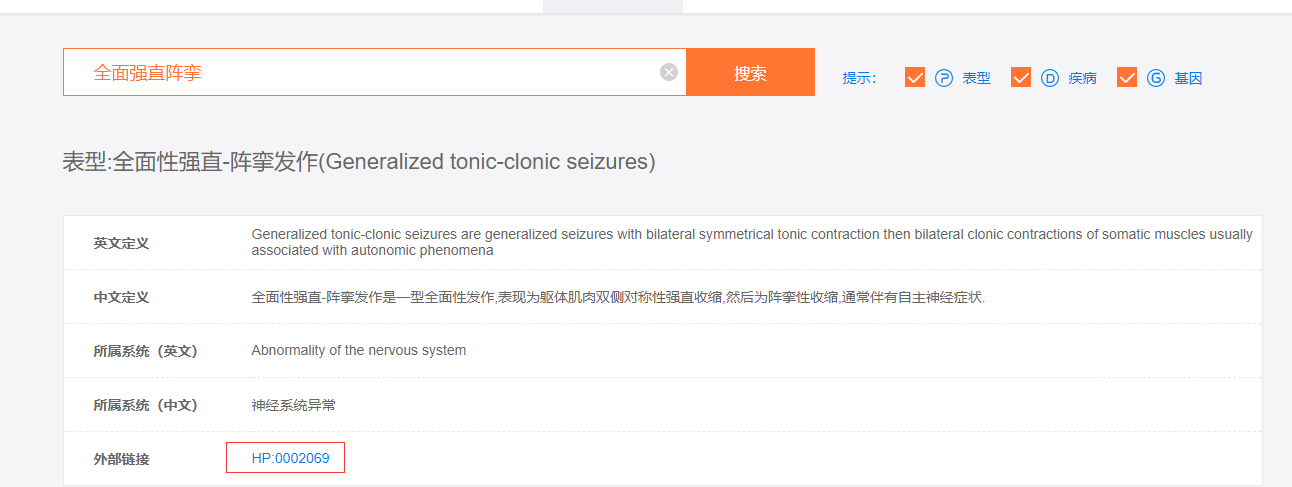 同理,能得到另外一个表型是HP:0002133。
同理,能得到另外一个表型是HP:0002133。
当然,如果表型越多,后面的分析也会越准确。但是前提是表型也要准确。
然后带上我们的vcf文件。
去准备配置文件。
配置文件的模板可以在exomiser的examples文件夹里面找到。
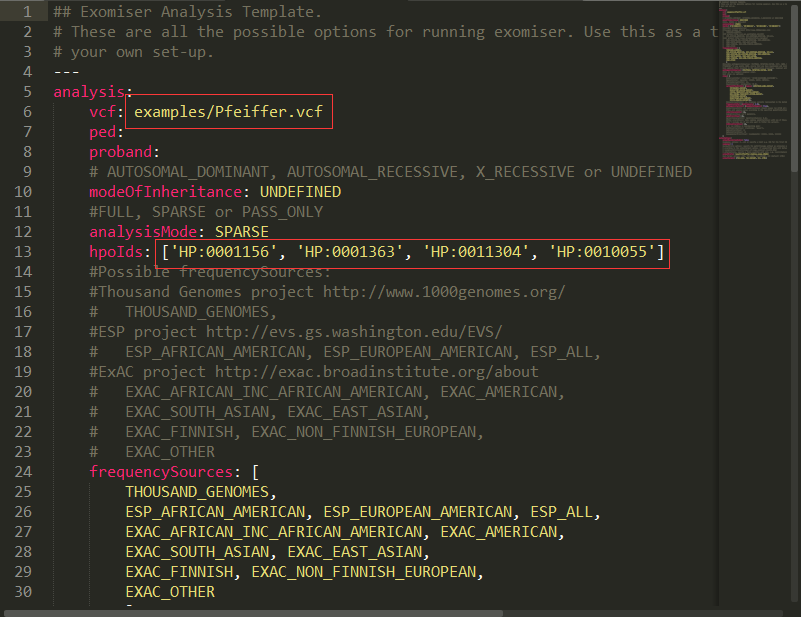 我们主要修改一下输入文件,相关表型,还有输出文件三个地方就ok。其实还有各种参数可以修改。exomiser的配置文件里面已经有了各个参数的注释,真是写得很好。
我们主要修改一下输入文件,相关表型,还有输出文件三个地方就ok。其实还有各种参数可以修改。exomiser的配置文件里面已经有了各个参数的注释,真是写得很好。
最后,打开你的终端,跑一下
java -Xms2g -Xmx4g -jar exomiser-cli-8.0.1.jar --analysis test.yml
跑完之后,在输出目录下就可以找到结果。
找到跑出来的html文件,用浏览器打开。
前面有各种信息。
我们可以主要关注一下Prioritised Genes这里。
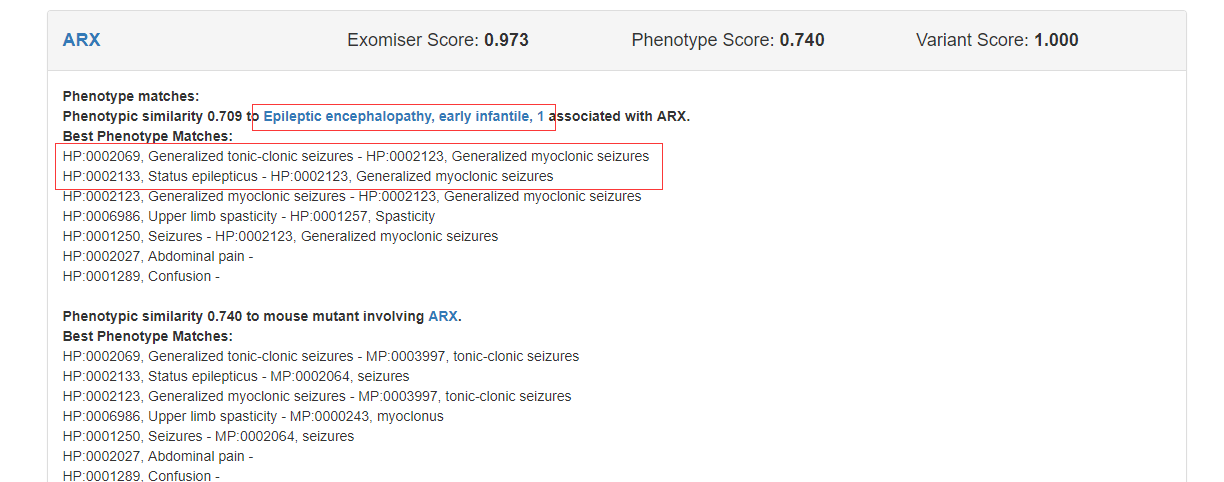
可以看到,它跑出来几个基因,其中,得分最高(exomiser score)的是ARX。这个基因包含了这两个表型。
接下来,我们就可以去用各种搜索工具各种数据库,去搜索一下这个基因和什么疾病有关。 然后看看这个疾病的临床症状,和患者的临床表现是否相符。
最后如果相符不要忘记去IGV看看这个位点的质量情况以及做一代测序验证。
Exomiser的作用不止这么一点,还能做家系分析之类的。这些多看看官方文档去学就好了。