要弄一个项目,首先要找到相关的panel。比如说,要做老年痴呆的项目,首先就应该去找关于阿尔兹海默病和额颞叶痴呆的相关基因位点。
刚好的是,发现一个统计了相关文献研究的网站。
然后选择基因可以看到这样的一个界面。
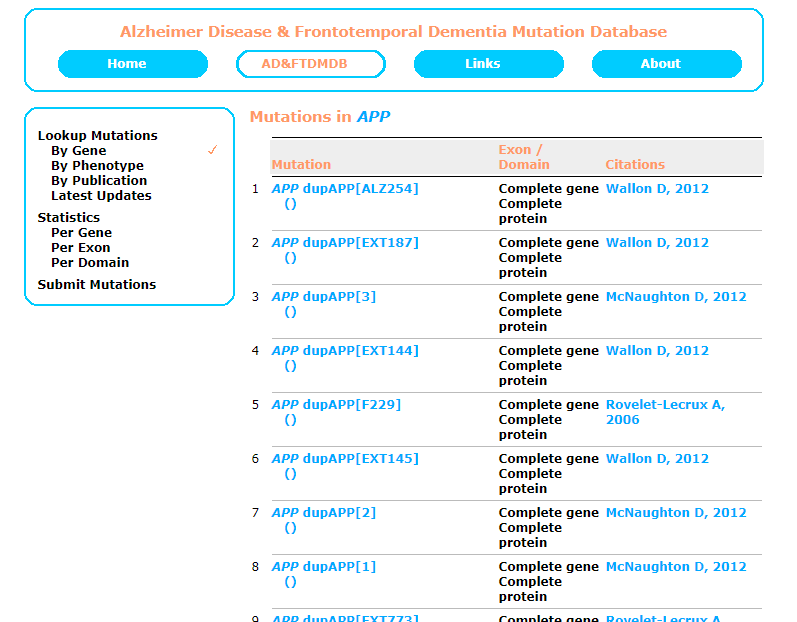
首选的弄下来的方法,当然是复制粘贴。
然而,复制粘贴格式会乱掉。 只好用脚本处理了。
我们可以找到这个网页的源代码。
点这里
下面是我用来处理的python脚本:
from bs4 import BeautifulSoup
inputFile = open('AD.html', 'r')
outputFile = open('result.txt', 'w')
soup = BeautifulSoup(inputFile, 'lxml')
gene = soup.select('p')
genes = []
for i in range(len(gene)):
if str(gene[i]).__contains__('span class'):
genes.append(gene[i])
genes.pop()
geneall = []
for m in range(len(genes)):
l = str(genes[m]).split('\n')
line = ''.join(l)
lines = ''.join(line.split())
genelist = lines.split('<b><i>')
del genelist[0]
genelist[-1] = genelist[-1].replace('</p>', '')
for n in range(len(genelist)):
geneall.append(genelist[n])
for x in geneall:
genename = x.split('</i></b>:')[0]
aachange = x.split('</i></b>:')[1]
aachanges = aachange.split('span')
del aachanges[0]
del aachanges[-1]
aachangess = ''.join(aachanges).split('>)(<')
for item in aachangess:
genedes = genename + '\t' + item.split('">')[0].split('"')[1] + '\t' + item.split('">')[1].split('<')[0]
outputFile.write(genedes + '\n')
outputFile.close()
inputFile.close()
print 'task done'
写的稍微乱了点不过可以跑通。
奉上处理之后的样子:
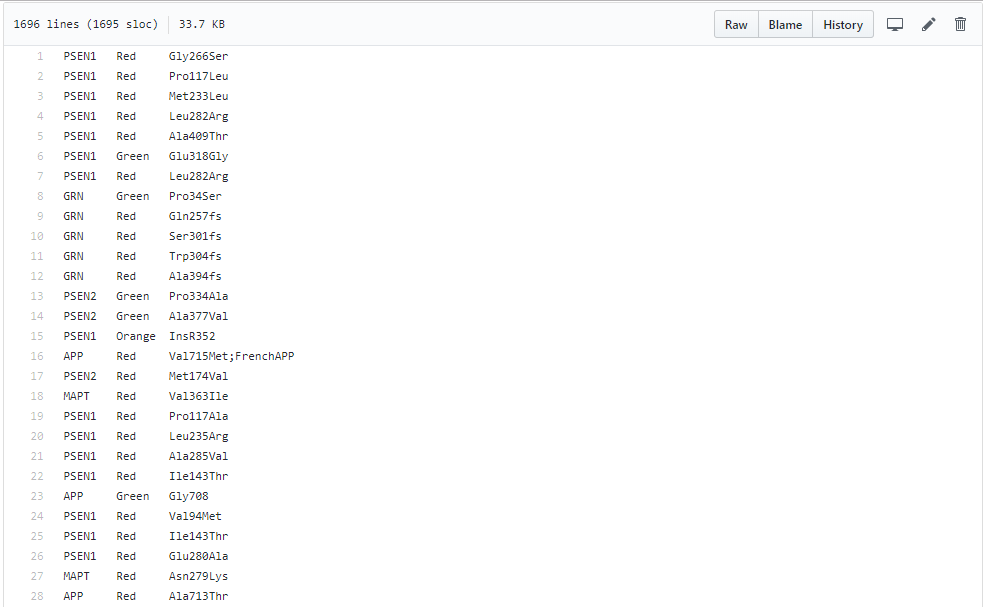
当然最后可以发现有一些位点重复了,这时候可以用万能的excel来进行处理了。毕竟用excel除重就2秒的事情。
最后这个其实并不是造出了一个panel,因为只有蛋白质变异的信息。
真正的panel需要的是位点信息。这个就要靠人工根据这些蛋白突变去查了。毕竟,没有免费的午餐。