最近在研究二代测序数据的HLA分型,找了一大堆分型软件。 很多安装过程和配置过程都很麻烦。
对比起来,这个是过程最简单的。
这是项目的页面seq2HLA
项目已经从bitbucket搬迁到了github了。
下载软件并解压
wget https://bitbucket.org/sebastian_boegel/seq2hla/get/seq2HLA_v2.2.tar.gz
tar -zxvf seq2HLA_v2.2.tar.gz
软件的运行需要bowtie,要先安装好并加载到环境变量中。 可以使用conda安装
conda install bowtie
也可以从官网安装。
可以参照软件自带的说明
python seq2HLA.py -h
如果数据是多条lane的数据,可以先合并起来。举个例子。
zcat read_L1_1.fq.gz read_L2_1.fq.gz > read_1.fq.gz
zcat read_L1_2.fq.gz read_L2_2.fq.gz > read_2.fq.gz
我们只需要使用一个简单命令就好了。数据需要的是双端的fq数据,支持gz压缩格式。
python seq2HLA -1 read_1.fq.gz -2 read_2.fq.gz -r output > output_summary.txt
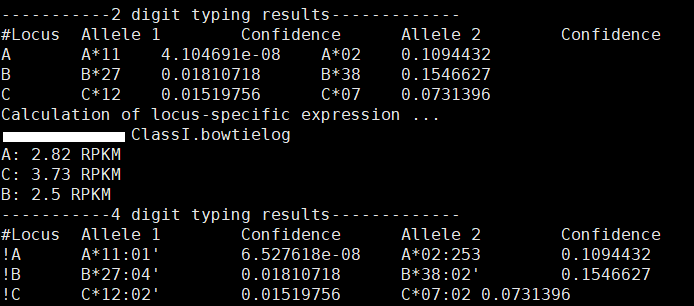
最后的结果是这样的: