DECoN是一款可以检测panel外显子缺失的R语言软件,使用的时候也是遇到一些问题,不过总体上还是挺好用的。DECoN可以在github下载。
爬坑1
软件依赖很多R包,理论上已经可以打包下载下来了,可能需要先来一下这一步
sh setup.sh
但是这一步我运行过后没有什么效果,运行的时候该报错还是报错,所以,建议是,把IdentifyFailures.R、ReadInBams.R、makeCNVcalls.R这三个脚本的第一行注释掉。然后运行的时候提示缺少什么R包再去手动安装。
source("https://bioconductor.org/biocLite.R")
biocLite("Biostrings")
biocLite("IRanges")
biocLite("Rsamtools")
biocLite("GenomicRanges")
biocLite("GenomicAlignments")
install.packages("reshape")
这里列一些需要的,还有ggplot2这种应该大家都会有了的。
爬坑2
软件用的时候直接使用的是”Rscript xxx.R”,但是不同环境下Rscript的默认载入包可能有点不同,我在运行的时候遇到说没有R.utils这个包。解决的方法是,指定需要载入的包,就是加上这一行。
Rscript --default-packages=methods,datasets,utils,grDevices,graphics,stats
流程
软件的输入实际上需要的是若干个bam文件(需要有对比),两个bed文件(其实一个就行但是我觉得两个比较好),参考基因组。
读入
从原始数据得到bam文件之后,读入bam,注意文件夹里除了bam文件外还需要bai索引
Rscript --default-packages=methods,datasets,utils,grDevices,graphics,stats \
ReadInBams.R --bams /path/to/bam-files/ \
--bed hg19.BRCA.bed \
--fasta hg19.fa \
--out results
这里的bed文件有4列,分别是chromosome、start、end、gene,不能有标题。会生成一个results.Rdata。
质检
这一步应该是说过不了质检的就算有结果都不能相信(?)。
Rscript --default-packages=methods,datasets,utils,grDevices,graphics,stats \
IdentifyFailures.R --Rdata results.Rdata \
--mincorr 0.98 --mincov 100 --exons hg19.BRCA.exons.bed \
--custom TRUE --out results.corr
这一步的bed文件又不一样,有5列,分别是Chr、Start、End、Gene、Custom.Exon,有标题。实际上就是可以自己定义哪个位置是哪个外显子。
最后就是检测
用的bed文件和质检用的一样,记得用了这种bed文件,custom要设置成true。
Rscript --default-packages=methods,datasets,utils,grDevices,graphics,stats \
makeCNVcalls.R --Rdata results.Rdata --transProb 0.01 \
--exons hg19.BRCA.exons.bed --custom TRUE --out results.CNV \
--plot All --plotFolder DECoNPlots
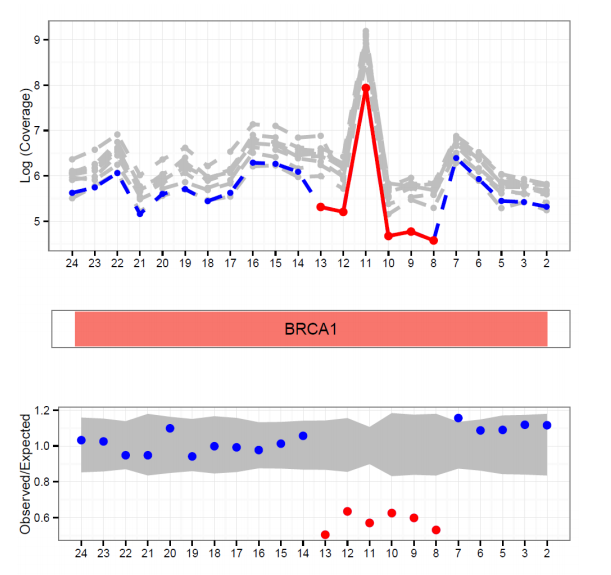
结果
xxx_all.txt和xxx_custom.txt文件都是汇总结果,但是custom文件是有我们自己定义的外显子信息的,所以看这个就行了。
然后图的话,像这样,每个阳性结果都会有一张图。