Personal Cancer Genome Reporter (PCGR),作者是挪威奥斯陆大学癌症研究所的。感觉是进行了大量的工作,把各个数据库以及软件搭建了在一起,并且最终生成了一个阅读性极高的html报告。
软件安装
软件的安装极其简单,以目前的稳定版本为例,软件基于python3,首先需要安装toml包
pip install toml
下载软件
wget https://github.com/sigven/pcgr/archive/v0.8.1.tar.gz
tar -zxvf v0.8.1.tar.gz
然后,下载数据包,数据包里面有tcga,civic,vep等多个癌症相关或软件依赖的数据库,大小约15G。还有一个问题,就是数据包存放地址在google drive,需要魔法才能下载。如果是使用魔法流量包的也需要考量流量是否充足以及节点是否稳定。
下载地址: grch37、 grch38 需要将这个解压到pcgr文件夹内。
接下来下载docker镜像(docker的安装和使用自行google),大小约5G
docker pull sigven/pcgr:0.8.1
作者大概是往docker中封装了一个注释的程序,然后通过python脚本来操作数据库与docker,所以只需要装了docker,pull下镜像即可,并不需要懂docker。
软件运行
软件的输入文件是vcf或者cnv结果。对于vcf文件来说,需要Filter列存在PASS的位点,因为软件大概只会使用PASS的位点进行分析。 按照GATK的mutect2流程来说,在最后做一步FilterMutectCalls就能从mutect2结果中输出PASS的结果。
python pcgr.py --input_vcf ~/pcgr-0.8.1/examples/tumor_sample.COAD.vcf.gz --input_cna ~/pcgr-0.8.1/examples/tumor_sample.COAD.cna.tsv --tumor_purity 0.9 --tumor_ploidy 2.0 ~/pcgr-0.8.1 ~/pcgr-0.8.1/examples grch37 ~/pcgr-0.8.1/examples/examples_COAD.toml tumor_sample.COAD
程序提供了两组测试数据可供测试。
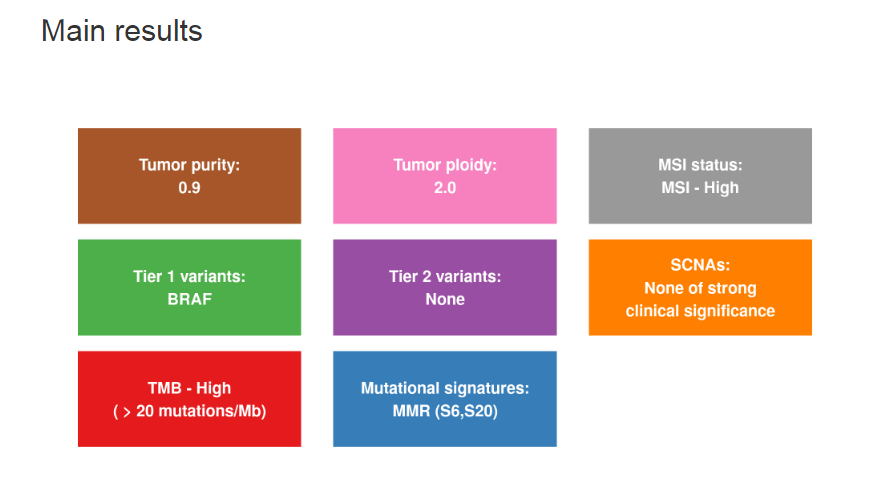
软件结果
软件会生成html结果。