乳腺癌有一个由21个基因组成,并且在指南里推荐了的复发评分模型。这个模型由Paik等提出。目前也有成熟的商品Oncotype DX。
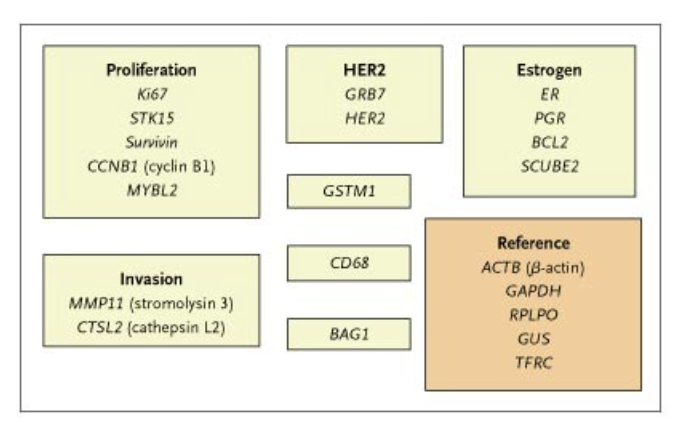
评分由以上6组基因组成,包括HER2组的GRB7、HER2;Proliferation组的Ki67、STK15、Survivin、CCNB1、MYBL2;Invasion组的MMP11、CTSL2;其他组的GSTM1、CD68、BAG1以及参考组的ACTB、GAPDH、RPLPO、GUS、TFRC。
各个基因使用qPCR获得CT值,然后用CT值计算获得最终的复发评分RS值。根据指南区分:低(RS < 26)、中(RS = 26到30)、高危RS(RS≥31)。另外,指南中对不同的年龄段也有了不同的RS划分。以下代码均为python。
测试用的CT值来源于迪安的一篇专利:
# RSu 9.70;RS 60.00
ctDict = {
"GRB7": 36.59, "HER2": 33.30,
"ER": 34.78, "PgR": 34.86, "BCL2": 35.90, "SCUBE2": 37.84,
"Survivin": 33.82, "Ki67": 35.00, "MYBL2": 34.76, "CCNB1": 33.21, "STK15": 35.42,
"CTSL2": 38.55, "MMP11": 31.80,
"CD68": 28.11, "GSTM1": 36.94, "BAG1": 34.83,
"ACTB": 24.74, "GAPDH": 27.23, "RPLP0": 28.71, "GUS": 36.33, "TFRC": 31.21
}
RS计算公式如下:
def BreastRS(geneDict):
# GRB7 group score = 0.9 × GRB7 + 0.1 × HER2 (if result < 8, then score is 8)
HER2_gs = 0.9 * geneDict["GRB7"] + 0.1 * geneDict["HER2"]
if HER2_gs < 8:
HER2_gs = 8
# ER group score = (0.8 × ER + 1.2 × PGR + BCL2 + SCUBE2) ÷ 4
ER_gs = (0.8 * geneDict["ER"] + 1.2 * geneDict["PgR"] + geneDict["BCL2"] + geneDict["SCUBE2"]) / 4
# proliferation group score = (Survivin + KI67 + MYBL2 + CCNB1 + STK15) ÷ 5 (if result < 6.5, then score is 6.5)
Prolifieration_gs = (geneDict["Survivin"] + geneDict["Ki67"] + geneDict["MYBL2"] + geneDict["CCNB1"] + geneDict["STK15"]) / 5
if Prolifieration_gs < 6.5:
Prolifieration_gs = 6.5
# invasion group score = (CTSL2 + MMP11) ÷ 2
Invasion_gs = (geneDict["CTSL2"] + geneDict["MMP11"]) / 2
# RSU = + 0.47 × GRB7 group score – 0.34 × ER group score + 1.04 × proliferation group score + 0.10 × invasion group score + 0.05 × CD68 – 0.08 × GSTM1 – 0.07 × BAG1
RSu = 0.47 * HER2_gs - 0.34 * ER_gs + 1.04 * Prolifieration_gs + 0.1 * Invasion_gs + 0.05 * geneDict["CD68"] - 0.08 * geneDict["GSTM1"] - 0.07 * geneDict["BAG1"]
# RS=0 if RSU<0; RS=20×(RSU–6.7) if 0≤RSU≤100; and RS=100 if RSU>100
if RSu <= 0:
RS = 0
elif RSu >= 100:
RS = 100
else:
RS = 20 * (RSu - 6.7)
return RS
对于以上计算RS的方法,不同公司引用时都是一样的,但是还需要对CT值进行标准化,而标准化的方式各有不同。
Paik文献中提到标准化的方式是把ΔCT值放缩到[0, 15]范围内,在github上找到一个oncotypedx的算法,基本参照了Paik文献的方式进行放缩,但是看了给的参考输入文件里并不是使用CT值作为原始输入(估计是相对量,这里也拿不到别人的对照组CT,因此无法使用2‐ΔΔCt 法来测试),另外这个算法也没有加入reference基因进行计算。
因此按照下面这个校正方法,并不能获得专利中的参考结果。
def scale_genefu(geneDict):
scaleDict = {}
geneList = [
"GRB7", "HER2",
"ER", "PgR", "BCL2", "SCUBE2",
"Survivin", "Ki67", "MYBL2", "CCNB1", "STK15",
"CTSL2", "MMP11",
"CD68", "GSTM1", "BAG1"
]
geneCT = []
for g in geneList:
geneCT.append(geneDict[g])
for g in geneList:
scaleCT = (geneDict[g] - min(geneCT)) / (max(geneCT) - min(geneCT)) * 15
scaleDict[g] = scaleCT
return scaleDict
scaleCTDict = scale_genefu(ctDict)
print(BreastRS(scaleCTDict))
根据观察 ,专利中的校正方法如下(可以理解为参考的初始量是10,每多一个CT就相当于初始量少1?):
def scale_d(geneDict):
referenceCT = [geneDict["ACTB"], geneDict["GAPDH"], geneDict["RPLP0"], geneDict["GUS"], geneDict["TFRC"]]
meanReferenceCT = sum(referenceCT) / len(referenceCT)
scaleDict = {}
for g in geneDict.keys():
deltaCT = geneDict[g] - meanReferenceCT
scaleCT = 10 - deltaCT
scaleDict[g] = scaleCT
return scaleDict
scaleCTDict = scale_d(ctDict)
print(BreastRS(scaleCTDict))