很忙很累还更新的我。
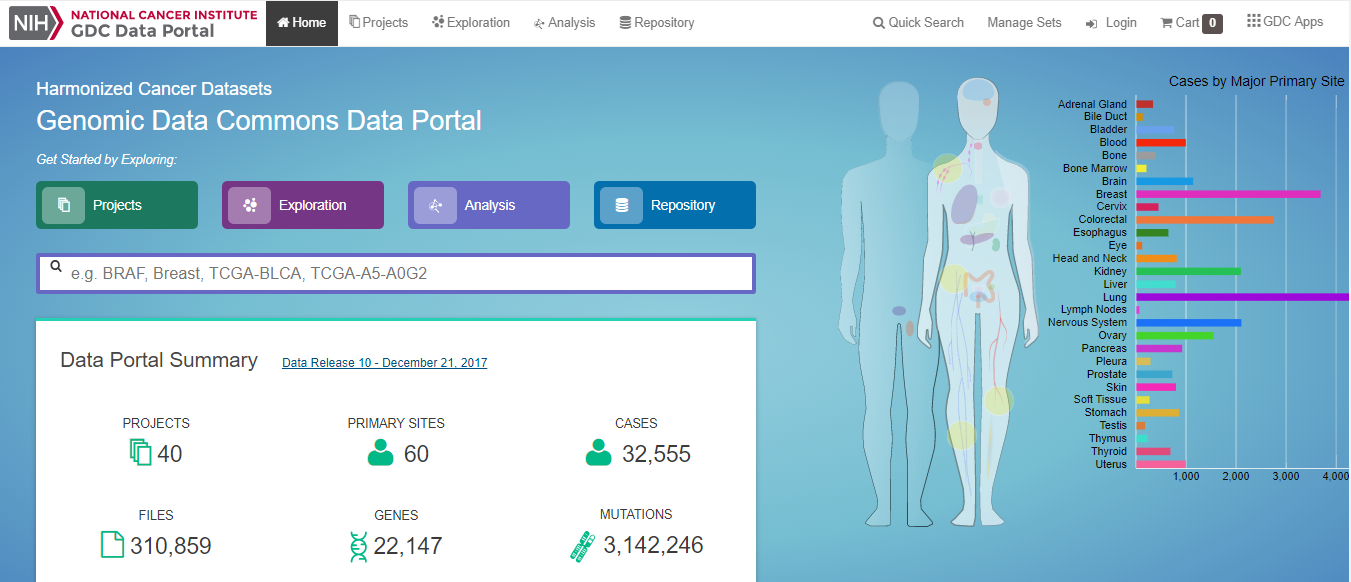
TCGA是一个癌症相关的数据库。可以弄到很多癌症的基因数据样本。 点点看TCGA。 进去之后,点右边那个蓝色的Launch Data Portal
可以看到样本量还是挺多的。
我们可以点右边的那个人的甲状腺(Thyroid)。 就进入了一个统计界面。 可以看到截至目前的相关突变位点有11128个。 单击Mutations,再点击JSON就可以把所有的突变位点下载下来。
然后我用下面这个脚本,提取出了所有的位点。
inputfile = open('Thyroid_mutation.json', 'r')
outputfile = open('results.txt', 'w')
for line in inputfile:
if line.startswith(' "genomic_dna_change":'):
a = line.split(' "genomic_dna_change": "')[1]
b = a.split('",')[0]
outputfile.write(b + '\n')
inputfile.close()
outputfile.close()
还是很简单的。
或者如果要的是所有的信息,每一个栏目统计好,可以这样:
import json
inputfile = open('Thyroid_mutation.json', 'r')
outputfile = open('results.txt', 'w')
analysisJson = json.load(inputfile)
for i in analysisJson:
consequence = i['consequence']
for j in consequence:
outList = []
transcript = j['transcript']
outDict = {'genomic_dna_change': '-', 'mutation_subtype': '-', 'gene': '-', 'aa_change': '-', 'impact': '-'}
outDict['genomic_dna_change'] = i['genomic_dna_change']
outDict['mutation_subtype'] = i['mutation_subtype']
outDict['gene'] = transcript['gene']['symbol']
if transcript['aa_change'] != None:
outDict['aa_change'] = transcript['aa_change']
if transcript.has_key('annotation'):
outDict['impact'] = transcript['annotation']['vep_impact']
outList.append(outDict['genomic_dna_change'])
outList.append(outDict['mutation_subtype'])
outList.append(outDict['gene'])
outList.append(outDict['aa_change'])
outList.append(outDict['impact'])
outStr = '\t'.join(outList) + '\n'
outputfile.write(outStr)
outputfile.close()
inputfile.close()