phyloseq是一个很好的用来对扩增子流程处理处理的OTU等数据,进行后续分析画图的包。 支持的上游软件包括了DADA2, UPARSE, QIIME, mothur, BIOM, PyroTagger, RDP。
首先在R中安装。
source("https://bioconductor.org/biocLite.R")
biocLite("phyloseq")
回过头来,上次说到的DADA2处理的数据。 实际上,其中的seqtab.nochim就是后续需要的otumat也就是OTU表格。 而taxa则是taxmat也就是taxa表格。
这些在phyloseq的文档里说的比较清晰。


先来把上次的结果导出。
write.csv(seqtab.nochim, "~/workspace/AMP/OTU/otumat.csv", quote=FALSE)
write.csv(taxa, "~/workspace/AMP/OTU/taxmat.csv", quote=FALSE)
然后开始画图的步骤。
引入包和主题
library(phyloseq)
library(ggplot2)
theme_set(theme_bw())
# 导入数据
otumat <- read.csv("~/workspace/AMP/OTU/otumax.csv", header=TRUE, quote="", row.names=1)
taxmat <- read.csv("~/workspace/AMP/OTU/taxmat.csv", header=TRUE, quote="", row.names=1)
# 转换为矩阵
otumat <- as.matrix(otumat)
taxmat <- as.matrix(taxmat)
# 读为otu和tax
# 这里由于DADA2输出的文件,样本名是行名(第一列),所以参数设置是FALSE。如果样本名是列名,则设置为TRUE
# 实际上,也可以直接默认TRUE,然后利用otu <- t(otu)这样改过来
otu <- otu_table(otumat, taxa_are_rows=FALSE)
tax <- tax_table(taxmat)
建立otu与tax的联系
physeq = phyloseq(otu, tax)
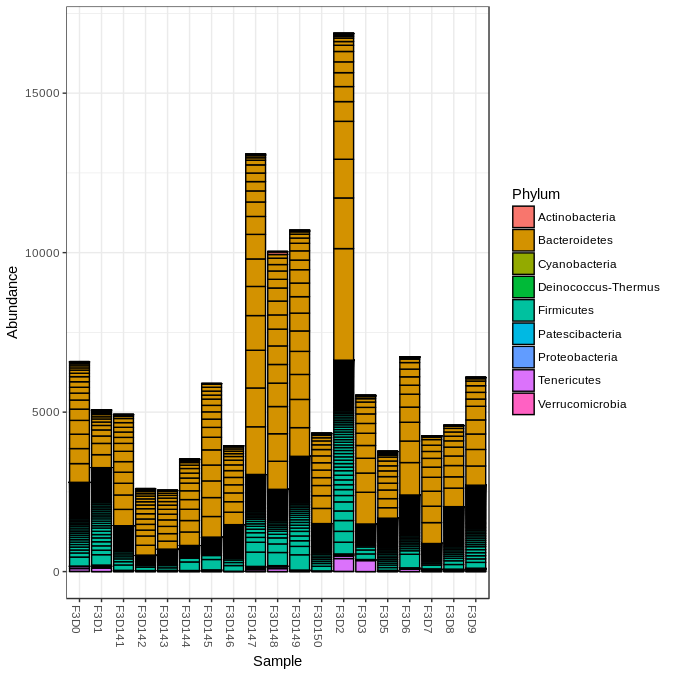
画物种丰度图
# 可以选择界门纲目科属种来画。
plot_bar(physeq, fill="Domain")
plot_bar(physeq, fill="Phylum")
plot_bar(physeq, fill="Class")
plot_bar(physeq, fill="Order")
plot_bar(physeq, fill="Family")
plot_bar(physeq, fill="Genus")
plot_bar(physeq, fill="Species")
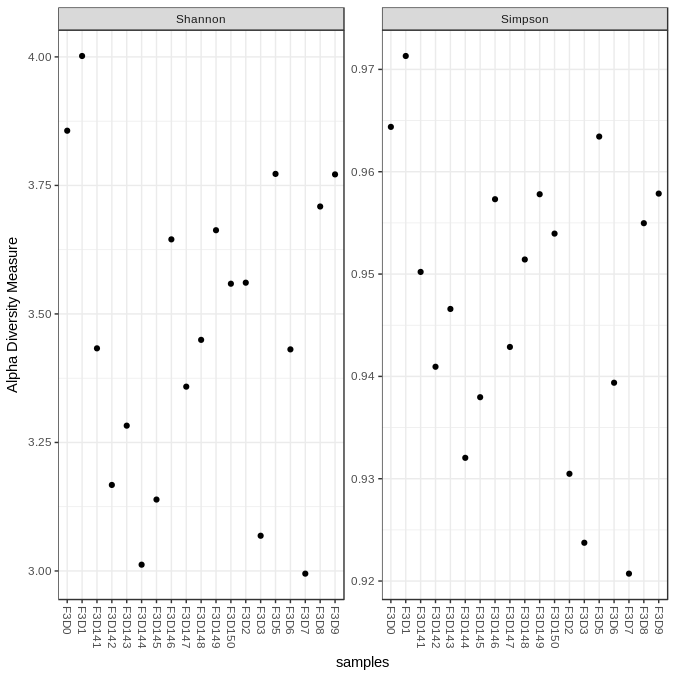
Alpha分析
# 可以把Shannon指数和Simpson指数分开画,也可以一起画
plot_richness(physeq, measures="Shannon")
plot_richness(physeq, measures="Simpson")
plot_richness(physeq, measures=c("Shannon", "Simpson"))
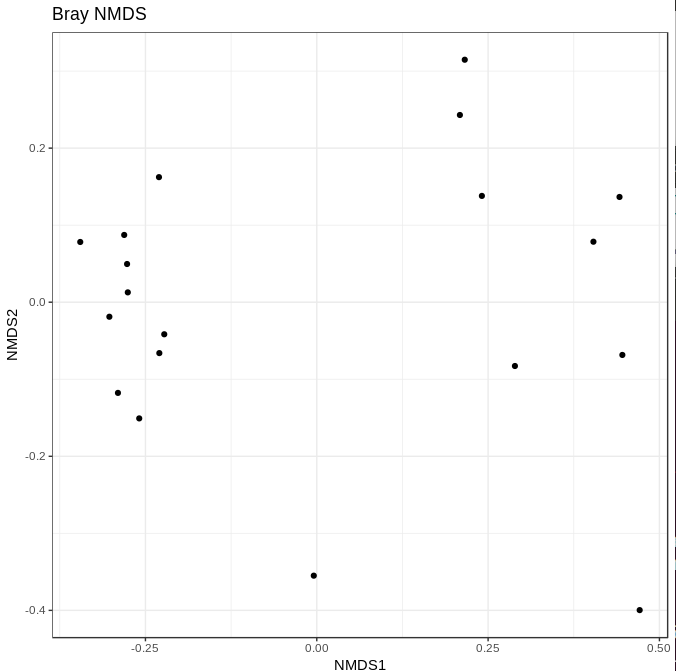
Beta分析
physeq.pop <- transform_sample_counts(physeq, function(otu) otu/sum(otu))
ord.nmds.bray <- ordinate(physeq.pop, method="NMDS", distance="bray")
plot_ordination(physeq.pop, ord.nmds.bray, title="Bray NMDS")